Fogalmak tisztázása
A szegmentáció egy kép több szegmensre vagy régióra történő felosztását jelenti bizonyos jellemzők, például szín, intenzitás, textúra vagy élek alapján. Célja, hogy a képeket ésszerűen részekre osztva egyszerűbbé tegye azoknak elemzés és megértés céljából történő ábrázolását. Az osztályozás során a fénykép pixelei vagy régiói bizonyos jellemzők alapján előre meghatározott osztályokba vannak sorolva. A klaszterezés lényege az adatok csoportosítása kinézetük alapján, előre meghatározott kategóriák nélkül. Gépi tanulási technika, amelynek az a célja, hogy természetes csoportokat találjon az osztályok vagy kategóriák előzetes ismerete nélkül.
CCA
Két lépésben egyszerűen szegmentálhatók bináris képek. Az első lépésben balról jobbra, fentről lefelé haladva az algoritmus minden fehér pixelnek megtekinti a szomszédjait. Ha azok mindegyike háttér, akkor új címkét kap az aktuális pixel. Ha a szomszédok nem hátterek és megegyeznek, akkor az ő címkéjüket kapja a vizsgált pixel. Ha különböző címkéjű fehér szomszédok vannak, a legkisebb címkét kell hozzárendelni, majd felírni az ekvivalenciát. A második lépés az ekvivalenciák alapján újracímkézi az összetartozó pixeleket.
Region growing
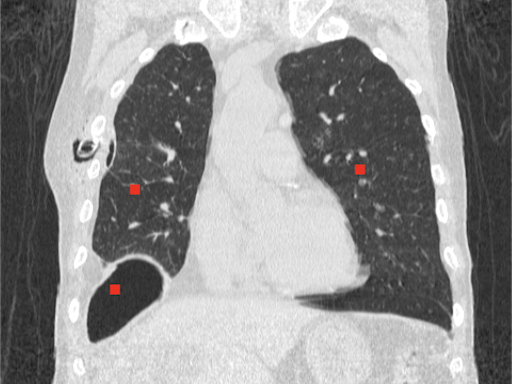
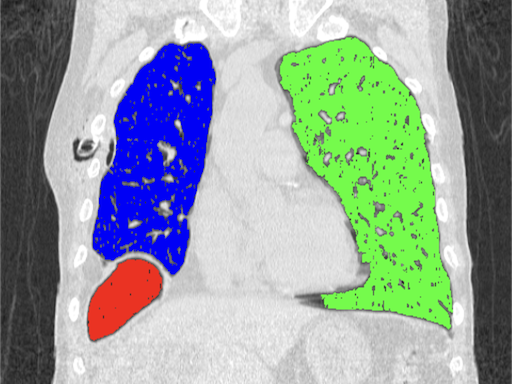
Az algoritmus a kijelölt pontokból indulva növeszt régiókat szomszédos pixelek hasonlósága alapján. Fontos megfelelően megadni a toleranciát, amely a megengedett százalékos eltérés szomszédok között. Lista készül a meglátogatandó szomszédokról, majd azoknak a szomszédai is felkerülnek a listára, a már meglátogatott pixelek pedig lekerülnek a listáról. Addig megy a folyamat, ameddig a lista teljesen ki nem ürül.
K-means
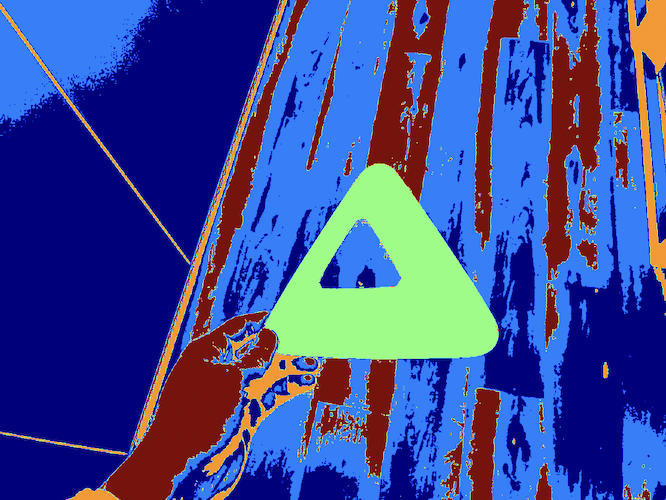
Egy iteratív klaszterezési módszer, amely képek szegmentálására is jól alkalmazható. Az algoritmus először véletlenszerűen kiválaszt K középpontot, ezután minden pixel hozzárendelődik a legközelebbi klaszterhez az intenzitásérték vagy egyéb jellemző alapján. Ezután az egyes klaszterekhez tartozó pixelek átlaga alapján új középpontokat kalkulál az algoritmus. Ez a folyamat ismétlődik addig, amíg a klaszterközéppontok már nem változnak jelentősen, vagy el nem ér egy előre meghatározott iterációs határt. A hasonló tulajdonságokkal rendelkező pixelek csoportokba rendeződnek, így elkülöníthetők az eltérő régiók.

További algoritmusok
Képek szegmentálása számos további módszerrel megvalósítható. A Watershed a bemenő adatot topográfiai térképként kezeli, a világosabb területeket magaslatokként, a sötétebbeket völgyekként értelmezi. Hasznos lehet összetett, egymással érintkező objektumok szegmentálásánál. A DBSCAN vagy Mean-Shift klaszterezés alapú, felügyelet nélküli gépi tanulási módszerek, nem igényelnek előre meghatározott klaszterszámot, így jól alkalmazhatók változatos adatstruktúrákra. A konvolúciós neurális hálózatok (CNN) is népszerűek különböző szegmentálási feladatok végrehajtására önvezető járművekben, egészségügyben és sok más területen.
Segédlet
scikit-learn telepítése: https://pypi.org/project/scikit-learn
Oválisok fénykép: Letöltés
Mellkasi cső fénykép: Letöltés
Bója fénykép: Letöltés
Forráskód: CCA
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
from skimage.measure import label
from skimage.color import label2rgb
# Open the image using PIL
image = Image.open("path-to-resources/ovals.jpeg").convert("L")
# Convert image to a NumPy array
data = np.array(image, dtype=np.uint8)
# Apply thresholding
threshold = 128
data = np.where(data > threshold, 0, 1)
# Label connected regions (CCA)
labels_from_image = label(data)
# Produce an image where labels are color-coded
image_label_overlay = label2rgb(labels_from_image, image=data, alpha=1.0)
# Display the image
plt.imshow(image_label_overlay)
plt.title("Center point")
plt.axis('off')
plt.show()
Forráskód: Region growing
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
# Open the image using PIL
# Image source: Case courtesy of Tariq Walizai, Radiopaedia.org, rID: 184833
# https://radiopaedia.org/cases/184833?lang=us
image = Image.open("path-to-resources/chest-tube.jpg").convert("RGB")
# Convert image to a NumPy array
data = np.array(image, dtype=np.uint8)
# Create a grayscale version, as well
grayscale_data = np.array(image.convert("L"), dtype=np.uint8)
seed_points = [[135,235],
[360,215],
[115,335]] # (X,Y) coordinates of points
color_values = [(255, 0, 0),
(0, 255, 0),
(0, 0, 255)] # (R, G, B)
# Create empty matrix to track visited points
visited_points = np.zeros((data.shape[1], data.shape[0]), dtype=np.uint8)
segmented_image = data.copy()
# Max. difference between pixels (%)
tolerance = 2
for p in range(len(seed_points)):
temp_list = [seed_points[p]]
while (len(temp_list) != 0):
curr_point = temp_list.pop() # Read then remove the last element of the list
x = curr_point[0]
y = curr_point[1]
im_val = grayscale_data[y][x] # Pixel value at the current point
visited_points[y][x] = 1 # Mark current point white
segmented_image[y][x] = color_values[p]
# Visit every neighbor (8c)
neighbors = [(x-1, y-1), (x-1, y+1), (x+1, y-1), (x+1, y+1), (x, y-1), (x, y+1), (x-1, y), (x+1, y)]
for i, j in neighbors:
if (0 <= j < data.shape[0] and 0 <= i < data.shape[1]): # The X and Y index are inside the image
neighbor_val = grayscale_data[j][i]
# Change the data type for proper subtraction
neighbor_val = neighbor_val.astype(np.int16)
# The point has not been visited and is similar to its neighbor
if (visited_points[j][i] == 0 and abs(neighbor_val - im_val) < tolerance / 100 * 255 ):
# LIFO method
temp_list.append([i,j])
# Display the image
plt.imshow(segmented_image)
# Mark seed points
plt.plot(seed_points[0][0], seed_points[0][1], "ow", markersize=5)
plt.plot(seed_points[1][0], seed_points[1][1], "ow", markersize=5)
plt.plot(seed_points[2][0], seed_points[2][1], "ow", markersize=5)
plt.title("Center point")
plt.axis('off')
plt.show()
Forráskód: K-means
from PIL import Image
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
# Open the image using PIL
image = Image.open("path-to-resources/cone.jpg")
# Convert image to a NumPy array
data = np.array(image, dtype=np.uint8)
# Get the dimensions of the image
image_h, image_w, _ = data.shape
# Reshape image data for k-means
pixels = data.reshape(-1, 3)
# Run the k-means algorithm
kmeans = KMeans(n_clusters = 4)
kmeans.fit(pixels)
# Get labels
labels = np.reshape(kmeans.labels_, (image_h, image_w))
# Display images
fig, ax = plt.subplots(1, 2, figsize=(12, 6))
ax[0].imshow(data)
ax[0].set_title("Original Image")
ax[0].axis("off")
ax[1].imshow(labels, cmap = "jet")
ax[1].set_title("Segmented Image")
ax[1].axis("off")
plt.show()